黑素瘤起源于神经嵴黑色素细胞,发病率在世界范围内逐步升高[1]。与其他皮肤恶性肿瘤相比,黑素瘤更罕见,但侵袭性更强,约73%的皮肤肿瘤相关死亡病例是黑素瘤导致的。黑素瘤因其突变基因[2]和较强的免疫原性可以进行靶向治疗和免疫治疗,但是亚洲人种靶向治疗和免疫治疗的效果劣于欧美高加索人种,因此,深入探索黑素瘤的发生发展机制,发现新的靶点对于疾病的治疗具有重要意义。
课题组前期研究[3-4]发现,E3泛素连接酶家族在黑素瘤的发生发展中起重要作用。环指蛋白157(RING finger protein 157, RNF157)具有E3泛素连接酶活性[5]。研究[6]发现,RNF157促进神经元细胞的存活,是关键调节因子;同时,RNF157可通过调节细胞周期和分裂在肿瘤增殖中发挥重要作用[6-8]。研究[9]显示,RNF157参与多条通路激活,是MAPK/ERK和PI3K/AKT信号通路的“连接点”,其中的ERK通路是调控细胞增殖相关的最经典通路之一。本研究旨在探讨RNF157在黑素瘤细胞中的生物学功能和潜在作用机制,并探索其与ERK通路之间的联系,为新的靶向治疗提供实验依据。
1 材料与方法 1.1 细胞培养和转染黑素瘤细胞株A2058和MV3购自中国科学院细胞库(上海)。A2058和MV3细胞均在添加10%胎牛血清(FBS;LONSERA)、链霉素(100 IU/mL)、青霉素(100 µg/mL)的改良Eagle培养基(DMEM;HyClone)中,置于37℃、5% CO2的恒温加湿培养箱中培养。A2058细胞和MV3细胞转染慢病毒或小干扰RNA(siRNA)分别构建RNF157过表达、敲减RNF157的细胞株。用于RNF157过表达的慢病毒构建物序列为plentia-ef1a-egfp-p2a-puro-cmv-RNF157-3flag。用于敲减RNF157的siRNA序列为CGAAGUGAGUGAUAACAGU。用于敲减小凹蛋白-1(caveolin-1,CAV1)的siRNA序列为CGAUACGUGGUCAAGAUU。通过实时荧光定量聚合酶链反应(qRT-PCR)和Western印迹实验评价转染效率。
1.2 qRT-PCR和Western印迹实验 1.2.1 qRT-PCR实验步骤参照既往文献[10]。使用RNA快速提取试剂盒(上海奕杉生物科技有限公司),按照试剂盒说明书提取细胞RNA,使用高容量cDNA反转录试剂盒(Takara)经反转录获得cDNA。分光光度仪测定cDNA浓度。以cDNA为模板,按SYBR荧光实时PCR试剂盒说明书配置10 μL反应体系,置于Q5实时荧光定量PCR仪,循环条件为94℃ 15 s, 60℃ 30 s, 72℃ 30 s。每个反应重复3次。采用GAPDH为内参,2﹣ΔΔCt法分析数据。RNF157引物合成于生工生物工程(上海)股份有限公司,序列为5'-GGTGGCTGAAGACGAAGTGAGTG-3'(F)、5'-AGAGGCAGAGGTGGCGACAG-3'(R)。GAPDH引物序列为5'-ACAACTTTGGTATCGTG-GAAGG-3'(F)、5'-GCCATCACGCCACAGTT-TC-3'(R)。
1.2.2 Western印迹实验实验步骤参照既往文献[3]。根据上样缓冲液(上海碧云天生物技术有限公司)说明书提取细胞总蛋白,BCA法测定蛋白浓度,水浴100℃加热10 min使蛋白变性。上样后进行SDS-PAGE电泳,转移至PVDF膜,5%胎牛血清封闭,4℃一抗孵育过夜,洗膜,室温二抗孵育2 h,使用ECL试剂发光。采用ImageJ软件(NIH公司, 美国)统计相对灰度值。实验使用一抗分别为:RNF157(WH0114804M1,Sigma公司)、DYKDDDDK Tag Recombinant Antibody(80010-1-RR,Proteintech公司)、GAPDH(AF5009,上海碧云天生物技术有限公司)、CAV1(16447-1-AP,上海碧云天生物技术有限公司)、p-ERK(4370,CST公司)、ERK(4695,CST公司)。
1.3 MTT实验及克隆形成实验 1.3.1 MTT实验实验步骤参照文献[11]。将细胞接种于96孔细胞培养板(1 000个/孔),加入完全培养基,重复3次。在指定时间点,每孔加入10 μL MTT,即3-(4, 5-二甲基噻唑-2)-2, 5-二苯基四氮唑溴盐溶液(5 mg/mL,上海碧云天生物技术有限公司),37℃孵育4 h。使用分光光度仪测定590 nm光密度(D)值。
1.3.2 克隆形成实验实验步骤参照既往文献[12]。将细胞接种于6孔板(1 000个/孔),加入完全培养基4 mL,置于37℃、5% CO2的恒温加湿培养箱中培养,每4 d更换培养基,10 d后4%多聚甲醛固定,结晶紫染色,于光学显微镜下拍照,使用ImageJ软件统计实验结果。
实验步骤参照既往文献[4]。4周龄无胸腺BALB/c裸鼠购于复旦大学附属中山医院动物实验中心,于SPF级动物房内饲养。分别向裸鼠腋下注射107个对照组或RNF157过表达黑素瘤细胞,4周后处死裸鼠,取出皮下瘤组织进行称重,并对瘤体分别切片进行Ki-67免疫组化染色实验。
1.5 免疫组化染色实验步骤参照既往文献[13]。4%多聚甲醛固定,梯度酒精脱水,二甲苯透明处理,梯度酒精水化,抗原修复,洗涤,灭活内源性酶,封闭液封闭,滴加一抗湿盒4℃过夜,洗涤,二抗室温孵育30 min,洗涤,滴加霉菌抗生物素-过氧化氢溶液室温10 min,洗涤,滴加DAB染色。
1.6 免疫共沉淀(co-immunoprecipitation, Co-IP)、蛋白质组学、泛素化组学、质谱分析实验步骤参照既往文献[14]。用RIPA裂解细胞,冰摇30 min, 12 000×g离心5 min,收集上清液。上清与一抗和Protein A/G磁珠在4℃孵育过夜。收集磁珠加入1×上样缓冲液煮沸10 min, Western印迹实验检测蛋白表达水平。扩增RNF157过表达组及对照组黑素瘤细胞株,于杭州景杰生物科技股份有限公司行蛋白质组学、泛素化组学及质谱分析。
1.7 统计学处理采用GraphPad Prism软件分析数据,实验数据以x±s表示,两组间比较采用非配对t检验,检验水准(α)为0.05。
2 结果 2.1 RNF157在黑素瘤组织中表达水平明显高于瘤旁组织利用课题组前期制备的黑素瘤组织及瘤旁组织芯片进行免疫组化染色,检测RNF157表达水平。结果显示,RNF157主要分布于黑素瘤组织的细胞质中,细胞核内也有少量分布(图 1A)。RNF157在黑素瘤组织中表达量高于瘤旁组织,差异有统计学意义(图 1B,P<0.01)。

|
| 图 1 黑素瘤组织与瘤旁组织中RNF157分布及表达水平 A:免疫组化染色显示RNF157在黑素瘤组织及瘤旁组织内的分布;B:黑素瘤组织及瘤旁组织的RNF157相对表达量。n=60,**P<0.01。 |
采用siRNA构建敲减RNF157的黑素瘤细胞系,选择转染效率更高的si3序列进行敲减细胞系的构建及扩增;采用含有flag标记的慢病毒载体构建RNF157过表达的黑素瘤细胞系。通过Western印迹实验及qRT-PCR实验验证转染效率。结果(图 2A)表明,MV3过表达组和A2058过表达组的RNF157的相对表达量显著高于对照组(P<0.000 1);si3转染效率最高,MV3 si3组和A2058 si3组的RNF157相对表达量显著低于对照组(P<0.01)。MTT实验(图 2B)、克隆形成实验(图 2C)结果表明,在黑素瘤细胞MV3及A2058中,RNF157过表达可显著促进黑素瘤细胞增殖,敲减RNF157则抑制黑素瘤细胞增殖,表明RNF157具有可以促进黑素瘤细胞增殖的作用。
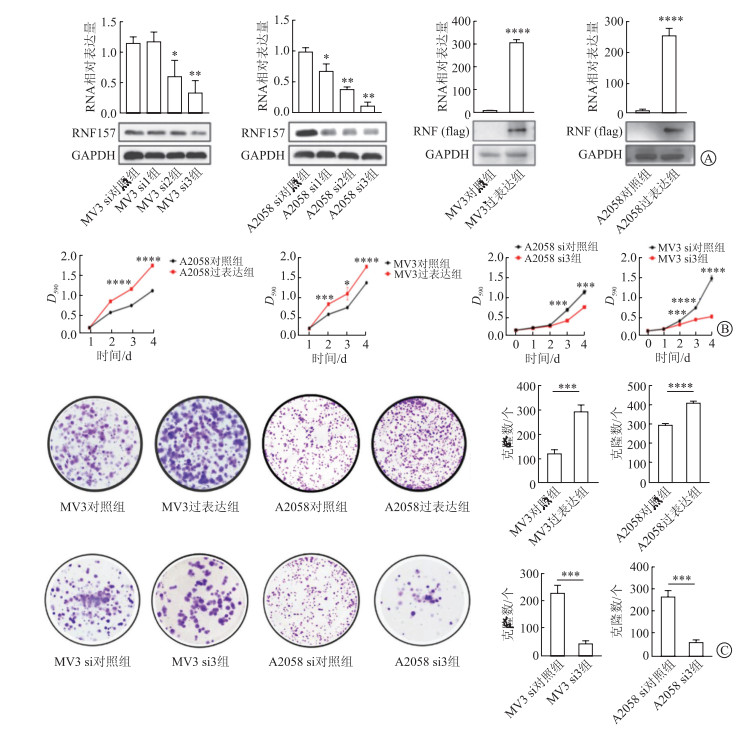
|
| 图 2 RNF157促进黑素瘤细胞增殖 A:qRT-PCR和Western印迹实验验证慢病毒和siRNA转染效率;B:MTT实验分析RNF157过表达和敲减RNF157的黑素瘤细胞的增殖能力;C:克隆形成实验分析RNF157过表达和敲减RNF157的黑素瘤细胞的增殖能力。n=3,x±s;*P<0.05,**P<0.01,***P<0.001,****P<0.000 1。 |
裸鼠皮下成瘤实验结果(图 3A)显示,过表达组的肿瘤重量明显大于对照组。免疫组织化学染色结果(图 3B)显示,过表达组皮下黑素瘤组织中Ki-67阳性率高于对照组,提示过表达RNF157可促进体内黑素瘤的生长。
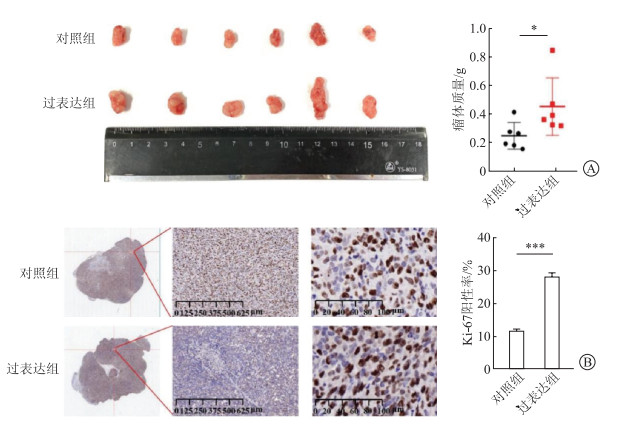
|
| 图 3 RNF157促进黑素瘤细胞体内增殖 A:对照组及过表达组黑素瘤瘤体大体观及重量;B:免疫组化染色比较过表达组及对照组Ki-67阳性率。n=6,x±s;*P<0.05,***P<0.001。 |
Western印迹实验结果(图 4)显示,在过表达RNF157的黑素瘤细胞内,磷酸化ERK(p-ERK)信号表达量增加,说明过表达RNF157促进ERK通路激活。

|
| 图 4 过表达RNF157促进黑素瘤细胞内ERK通路激活 RNF157:环指蛋白157;p-ERK:磷酸化ERK;t-ERK:总ERK。n=3,x±s;**P<0.01,***P<0.001。 |
对RNA157过表达组及对照组A2058细胞进行蛋白质组学及泛素化组学研究,结果(图 5A)显示,在过表达组中,共有286个上调蛋白,230个下调蛋白;231个上调的泛素化位点,264个下调的泛素化位点。根据RNF157的作用机制选择表达量下调、泛素化位点上调的蛋白质。同时,用Flag抗体对RNA157过表达A2058细胞进行Co-IP及质谱分析,以明确RNF157的相互作用蛋白。将蛋白质组学及泛素化组学得出的数据取交集制作韦恩图(图 5B),以获取RNF157泛素化调控底物蛋白。底物蛋白需满足以下条件:(1)过表达RNF157能增加其泛素化水平;(2)过表达RNF157降低其蛋白表达水平;(3)与RNF157存在相互作用。共筛选出2个蛋白,即CAV1及CAVIN1。为证明CAV1与RNF157的相互作用,进一步行Co-IP实验,结果(图 5C)表明,RNF157与CAV1在黑素瘤细胞A2058、MV3中存在相互作用。Western印迹实验结果(图 5D)显示,过表达RNF157可引起黑素瘤细胞内CAV1含量降低。
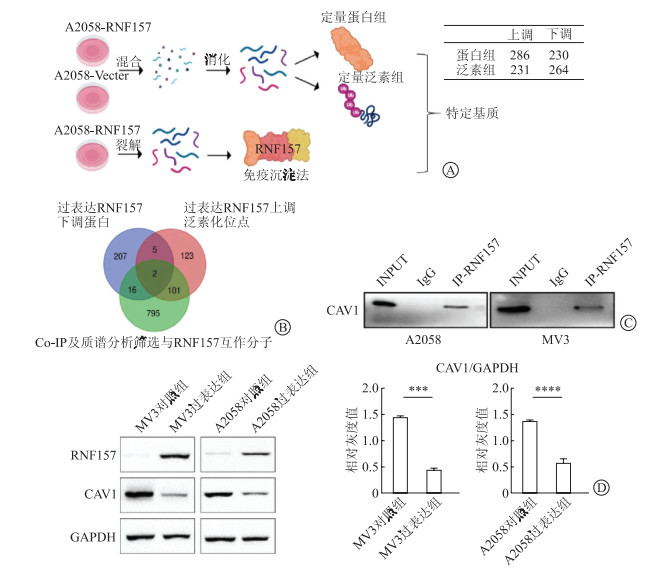
|
| 图 5 RNF157与CAV1存在相互作用 A、B:基于蛋白质组学、泛素化组学获取过表达RNF157组黑素瘤细胞内泛素化位点上调和表达量下调的蛋白质,Co-IP结合质谱分析筛选与RNF157相互作用的蛋白,取交集筛选目标蛋白;C:Co-IP验证RNF157与CAV1相互作用;D:Western印迹实验检测RNF157与CAV1表达量之间的关系。RNF157:环指蛋白157;CAV1:小凹蛋白-1。n=3,x±s;***P<0.001,****P<0.000 1。 |
采用回复实验进一步探究CAV1在RNF157介导的黑素瘤增殖中的作用。结果(图 6A)显示,敲减RNF157后克隆集落数量明显减少,而敲减CAV1可以使得敲减RNF157后的克隆集落数量部分回升。Western印迹实验结果(图 6B)显示,RNF157可以负向调控CAV1含量,而敲减CAV1并不能逆转RNF157的表达。同时,敲减RNF157可引起p-ERK表达水平降低,而进一步敲减CAV1后,这种抑制作用被部分抵消,提示敲减CAV1可以部分逆转敲减RNF157带来的黑素瘤细胞增长抑制。
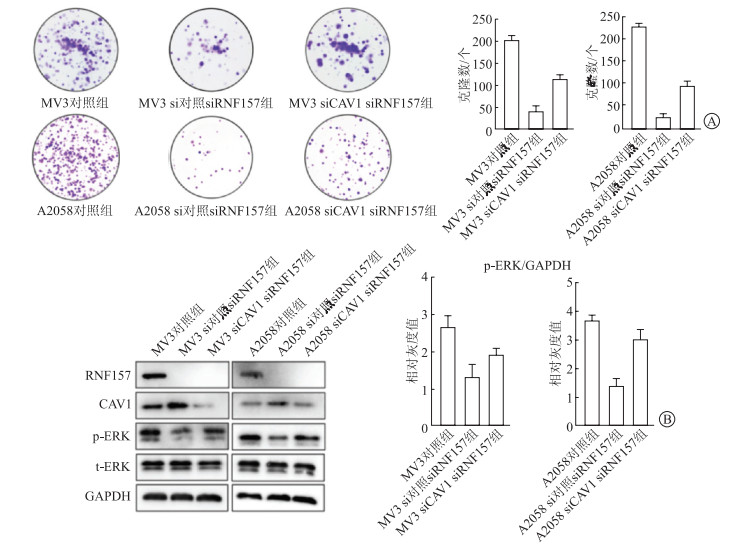
|
| 图 6 回复实验证实CAV1可部分逆转敲减RNF157对黑素瘤细胞增殖的抑制 A:克隆形成实验检测细胞增殖能力;B:Western印迹实验检测RNF157、CAV1、p-ERK、t-ERK及信号通路表达量变化。RNF157:环指蛋白157;CAV1:小凹蛋白-1;p-ERK:磷酸化ERK;t-ERK:总ERK。n=3,x±s;*P<0.05,**P<0.01,***P<0.001,****P<0.000 1。 |
本研究通过构建RNF157过表达黑素瘤细胞系和敲减RNF157黑素瘤细胞系,采用多种体内外表型实验证实了RNF157在黑素瘤中表达上调,RNF157过表达可以促进黑素瘤细胞的增殖。研究[6-7]表明,RNF157的升高可促进神经元细胞的存活和增殖。然而,本研究首次发现RNF157在黑素瘤细胞的增殖中起到促进作用,并尝试探究了其可能的作用机制。
有文献[7]报道,RNF157可能参与多种通路的调控,如PI3K/MAPK;然而,目前还缺乏将RNF157与这些通路联系起来的研究。本研究结果显示,RNF157的上调可促进A2058和MV3黑素瘤细胞内ERK通路的激活。ERK通路是MAPK通路的最后一个组成部分,是MAPK级联中研究最充分的通路,与细胞增殖、生长、分化等生物学过程密切相关。本研究结果表明,在过表达RNF157的黑素瘤细胞内,p-ERK信号表达量增加,说明RNF157可能通过参与调控ERK通路来影响黑素瘤细胞的增殖。
RNF157是一种E3泛素连接酶,通过参与体内泛素化过程发挥作用。RNF157促进泛素分子与底物蛋白结合,引起底物蛋白分子通过泛素-蛋白酶体途径降解[5, 15-16]。提示RNF157可能不直接参与ERK信号通路的调控,但通过调节ERK通路中涉及的蛋白质含量而间接产生影响。本研究利用蛋白质组学、泛素化组学、质谱分析和Co-IP筛选出RNF157的相互作用分子CAV1。CAV1与某些肿瘤的发生发展密切相关,然而它的作用在不同的肿瘤类型中有很大的不同。有文献指出,CAV1具有肿瘤抑制因子和肿瘤启动因子的双重作用,这取决于肿瘤类型[16-17]。有研究指出CAV1表现出肿瘤抑制活性[18-19],同时有研究报道CAV1可抑制肿瘤细胞ERK信号通路[20]。本研究发现,过表达RNF157可负向调节CAV1在黑素瘤细胞中的含量,促进ERK通路激活,促进黑素瘤细胞增殖。回复实验证实敲减CAV1可部分逆转敲减RNF157对黑素瘤细胞增殖的抑制作用。此结果提示,RNF157可与黑素瘤细胞内的CAV1结合,引起CAV1表达降低;RNF157过表达可促进p-ERK表达,引起ERK通路激活上调,进而引起黑素瘤细胞增殖增加。同时,研究[21]指出,ERK通路被抑制后可以检测到CAV1的表达,提示CAV1与ERK通路之间可能存在相互调节。
本研究存在一定局限性。首先,由于RNF157分子相关抗体选择有限,功能尚不齐全,部分实验如Co-IP有待进一步完善,使研究结果更为全面严谨;其次,CAV1对黑素瘤细胞增殖能力的影响及机制有待未来进行更加深入的研究。
综上所述,RNF157可促进黑素瘤细胞的增殖;在黑素瘤细胞中,RNF157可能通过结合并下调CAV1表达水平,参与细胞内ERK通路激活。本研究为RNF157在黑素瘤细胞增殖能力调控中的作用提供了证据,补充完善了课题组关于E3泛素连接酶家族在黑素瘤中作用机制的研究,为进一步探究黑素瘤的分子机制和设计靶向治疗方法提供了新的研究方向和基础。
利益冲突:所有作者声明不存在利益冲突。
| [1] |
AUBUCHON M M, BOLT L J, JANSSEN-HEIJNEN M L, et al. Epidemiology, management and survival outcomes of primary cutaneous melanoma: a ten-year overview[J]. Acta Chir Belg, 2017, 117(1): 29-35.
[DOI]
|
| [2] |
FIDALGO F, GOMES E E, MOREDO FACURE L, et al. Association of melanoma with intraepithelial neoplasia of the pancreas in three patients[J]. Exp Mol Pathol, 2014, 97(1): 144-147.
[DOI]
|
| [3] |
WEI C Y, WANG L, ZHU M X, et al. TRIM44 activates the AKT/mTOR signal pathway to induce melanoma progression by stabilizing TLR4[J]. J Exp Clin Cancer Res, 2019, 38(1): 137.
[DOI]
|
| [4] |
WEI C Y, ZHU M X, YANG Y W, et al. Downregulation of RNF128 activates Wnt/β-catenin signaling to induce cellular EMT and stemness via CD44 and CTTN ubiquitination in melanoma[J]. J Hematol Oncol, 2019, 12(1): 21.
[DOI]
|
| [5] |
SULLIVAN J A, SHIRASU K, DENG X W. The diverse roles of ubiquitin and the 26S proteasome in the life of plants[J]. Nat Rev Genet, 2003, 4(12): 948-958.
|
| [6] |
MATZ A, LEE S J, SCHWEDHELM-DOMEYER N, et al. Regulation of neuronal survival and morphology by the E3 ubiquitin ligase RNF157[J]. Cell Death Differ, 2015, 22(4): 626-642.
[DOI]
|
| [7] |
DOGAN T, GNAD F, CHAN J, et al. Role of the E3 ubiquitin ligase RNF157 as a novel downstream effector linking PI3K and MAPK signaling pathways to the cell cycle[J]. J Biol Chem, 2017, 292(35): 14311-14324.
[DOI]
|
| [8] |
KOSACKA J, NOWICKI M, PAESCHKE S, et al. Up-regulated autophagy: as a protective factor in adipose tissue of WOKW rats with metabolic syndrome[J]. Diabetol Metab Syndr, 2018, 10: 13.
[DOI]
|
| [9] |
CODENOTTI S, FAGGI F, RONCA R, et al. Caveolin-1 enhances metastasis formation in a human model of embryonal rhabdomyosarcoma through Erk signaling cooperation[J]. Cancer Lett, 2019, 449: 135-144.
[DOI]
|
| [10] |
KE A W, SHI G M, ZHOU J, et al. CD151 amplifies signaling by integrin α6β1 to PI3K and induces the epithelial-mesenchymal transition in HCC cells[J]. Gastroenterology, 2011, 140(5): 1629-1641. e15.
[DOI]
|
| [11] |
KUMAR P, NAGARAJAN A, UCHIL P D. Analysis of cell viability by the MTT assay[J]. Cold Spring Harb Protoc, 2018, 2018(6).
|
| [12] |
CAI J B, SHI G M, DONG Z R, et al. Ubiquitin-specific protease 7 accelerates p14(ARF) degradation by deubiquitinating thyroid hormone receptor-interacting protein 12 and promotes hepatocellular carcinoma progression[J]. Hepatology, 2015, 61(5): 1603-1614.
[DOI]
|
| [13] |
WANG L, SONG D L, WEI C Y, et al. Telocytes inhibited inflammatory factor expression and enhanced cell migration in LPS-induced skin wound healing models in vitro and in vivo[J]. J Transl Med, 2020, 18(1): 60.
[DOI]
|
| [14] |
CUI H R, WANG Q, LEI Z C, et al. DTL promotes cancer progression by PDCD4 ubiquitin-dependent degradation[J]. J Exp Clin Cancer Res, 2019, 38(1): 350.
[DOI]
|
| [15] |
SENFT D, QI J F, RONAI Z A. Ubiquitin ligases in oncogenic transformation and cancer therapy[J]. Nat Rev Cancer, 2018, 18(2): 69-88.
[DOI]
|
| [16] |
NWOSU Z C, EBERT M P, DOOLEY S, et al. Caveolin-1 in the regulation of cell metabolism: a cancer perspective[J]. Mol Cancer, 2016, 15(1): 71.
[DOI]
|
| [17] |
GUPTA R, TOUFAILY C, ANNABI B. Caveolin and cavin family members: dual roles in cancer[J]. Biochimie, 2014, 107(Pt B): 188-202.
|
| [18] |
YIN D D, QIU J Y, HU S X, et al. CAV1 is a prognostic predictor for patients with idiopathic pulmonary fibrosis and lung cancer[J]. J Biosci, 2022, 47: 13.
[DOI]
|
| [19] |
DÍAZ M I, DÍAZ P, BENNETT J C, et al. Caveolin-1 suppresses tumor formation through the inhibition of the unfolded protein response[J]. Cell Death Dis, 2020, 11(8): 648.
[DOI]
|
| [20] |
BERNASSOLA F, CHILLEMI G, MELINO G. HECT-type E3 ubiquitin ligases in cancer[J]. Trends Biochem Sci, 2019, 44(12): 1057-1075.
[DOI]
|
| [21] |
COHEN A W, PARK D S, WOODMAN S E, et al. Caveolin-1 null mice develop cardiac hypertrophy with hyperactivation of p42/44 MAP kinase in cardiac fibroblasts[J]. Am J Physiol Cell Physiol, 2003, 284(2): C457-C474.
[DOI]
|
 2023, Vol. 30
2023, Vol. 30


