慢性阻塞性肺疾病(chronic obstructive pulmonary disease, COPD)是一种以持续的呼吸道症状和气流受限为主要特征的常见呼吸系统疾病,目前全球范围内致死率仅次于缺血性心脏病和脑卒中[1],预计未来40年患病率将不断增加[2]。烟草暴露、空气污染、遗传因素和微生物组感染是其常见的致病因素,其中烟草暴露是最主要的危险因素[3]。
随着高通量16S rRNA测序技术的发展,人的肺部被发现存在丰富的微生物群落[4]。而多种肺部疾病的发生发展与肺部菌群的改变密切相关,如肺囊性纤维化[5-6]、重度哮喘[7]及COPD[8]。Haldar等[8]发现,健康受试者与COPD受试者的痰液微生物组存在显著差异,其中γ-变形菌纲与COPD恶化相关。但是,目前仍缺乏COPD患者肺实质微生物组相关研究。
胶质瘤发病相关蛋白1(GLI pathogenesis-related 1, GLIPR1)在人胶质母细胞瘤中高表达,可能是一种促癌蛋白[9]。课题组前期研究[10]发现,敲除GLIPR1基因能够通过调控PLAU/EGFR信号通路促进细胞焦亡,从而使小鼠香烟烟雾暴露后肺部炎症因子白介素(IL)-6、IL-1β释放增加,加重小鼠肺部损伤与气道重塑。而既往研究[11-12]发现,肺部菌群能调节哮喘、肺移植患者肺部炎症因子的分泌。因此,GLIPR1可能通过调节肺部菌群影响COPD进展。本研究通过16S rRNA测序技术分析GLIPR1基因对香烟烟雾暴露诱导的小鼠COPD模型肺部菌群的影响。
1 材料与方法 1.1 实验动物C57BL/6野生型雄鼠(8周龄)购于上海斯莱克实验动物有限责任公司。GLIPR1基因敲除鼠由上海南方模式生物科技股份有限公司采用CRISPR/Cas9技术,通过非同源重组修复引入突变方式获得。具体步骤:通过体外转录获得Cas9 mRNA和gRNA;将Cas9 mRNA和gRNA显微注射到C57BL/6J小鼠的受精卵中,经PCR产物测序确认获得GLIPR1基因蛋白读码框移码的F0代小鼠;F0代小鼠与C57BL/6小鼠交配获得F1代小鼠;F1代小鼠继续进行繁育,获得纯合的全身性GLIPR1基因敲除鼠。动物饲养于复旦大学附属中山医院实验动物中心。在饲养过程中,小鼠自由饮水并获取食物,每日接受12 h光照。动物实验获得复旦大学附属中山医院实验动物伦理委员会审批(2022-032)。
1.2 实验分组将小鼠随机分为野生型对照组(WT组)、野生型香烟烟雾暴露组(WT_SMOKE组)、GLIPR1基因敲除对照组(GLIPR1组)和GLIPR1基因敲除香烟烟雾暴露组(GLIPR1_SMOKE组),每组4~6只。WT_SMOKE组与GLIPR1_SMOKE组小鼠暴露于45 cm×25 cm×25 cm亚克力箱中,被动吸入二手烟(大前门:每支焦油11 mg、烟气烟碱0.8 mg、一氧化碳13 mg;上海烟草集团有限责任公司),每周5 d,每天20支香烟,持续3个月。造模结束后,小鼠右上肺组织苏木精-伊红(H-E)染色显示,气道显著增厚,肺泡间隔严重破坏,出现典型的COPD气道重塑与肺气肿表型,表明造模成功[10]。
1.3 小鼠血液及肺组织获取小鼠香烟烟雾暴露3个月后,用1.25%三溴乙醇(阿佛丁,20 mL/kg)腹腔注射麻醉小鼠,眼球取血,充分暴露小鼠胸腔,取肺。将小鼠右中肺取出置于冻存管中,﹣80℃保存,备用。
1.4 DNA抽提、建库使用DNA抽提试剂盒(DNeasy PowerSoil Kit QIAGEN 12888)提取样本基因组DNA,通过琼脂糖凝胶电泳和NanoDrop2000检测DNA浓度。以基因组DNA为模板,用带barcode的特异引物对细菌的16S V3~4区进行PCR(Tks Gflex DNA Polymerase),确保扩增效率和准确性。PCR产物通过电泳检测,检测后用磁珠纯化;纯化后作为模板再次进行PCR扩增,电泳检测,磁珠纯化,对扩增产物进行定量。
1.5 高通量测序通过Illumina MiSeq高通量二代测序平台获得肺组织样本16S rDNA序列信息。通过Trimmomatic软件对16S rDNA原始双端序列进行去杂,去杂后用FLASH软件进行拼接。经质控获得优质序列,通过Vsearch软件,将相似度≥97%的序列归为一个操作分类单元(OTU)。用QIIME软件包挑选出各个OTU的代表序列,并将所有代表序列与Silva数据库(V123)进行比对注释。
1.6 肺部菌群多样性分析以α多样性表示肺部菌群丰富度与多样性,主要指标包括Chao1指数(物种丰富度)、Shannon指数与Simpson指数(物种多样性)。Chao1指数和Shannon指数越高、Simpson指数越低,表示物种丰富度与多样性越高。通过主坐标分析(principal coordinate analysis, PCoA)反映各组间肺部菌群构成差异,即β多样性。通过线性判别分析效应大小(linear discriminant analysis effect size, LEfSe)分析识别各组潜在标志菌群。
1.7 统计学处理采用R软件程序包(V4.1.13)进行数据分析。计量资料以x±s表示,多组间比较采用单因素方差分析(ANOVA)或Kruskal Wallis H检验,两组间比较采用t检验或Wilcoxon检验。通过Tukey检验计算OTU的相对丰度差异。通过Galaxy/Hutlab在线平台(http://huttenhower.sph.harvard.edu/galaxy)进行线性判别分析(LDA)和LEfSe分析。检验水准(α)为0.05。
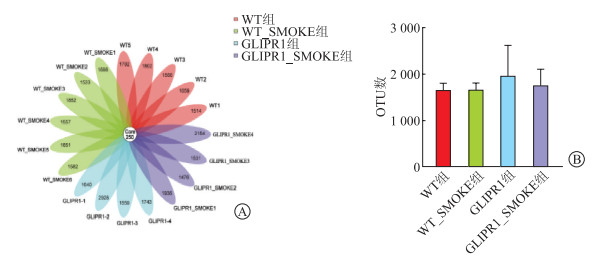
2 结果 2.1 肺部菌群物种丰富度 2.1.1 总体OTU通过小鼠肺组织16S rRNA测序,共获得10 332个OTU,稀疏曲线表明,单个样本的OTU数已达饱和,很难通过扩大测序数量获得更大的OTU数。有250个OTU序列共存于4组样本中,4组OTU总数差异无统计学意义(图 1)。
|
| 图 1 物种分析注释OTU数 A:OTU分布韦恩图;B:4组小鼠肺组织OTU总数比较。OTU:操作分类单元。 |
结果(图 2A、图 2B)显示:拟杆菌门、厚壁菌门、变形菌门和放线菌门在所有小鼠肺组织中为优势菌门。WT组小鼠前4位的优势菌属为拟杆菌属、柔嫩梭菌属、普雷沃氏菌属和乳杆菌属(Lactobacillus),占比分别为12.605%、2.004%、3.467%和3.104%。与WT组相比,拟杆菌属和柔嫩梭菌属丰度在WT_SMOKE组增加,分别为15.078%、2.885%,在GLIPR1_SMOKE组进一步增加,分别为19.742%、4.237%;乳杆菌属丰度在WT_SMOKE组减小(2.156%),在GLIPR1_SMOKE组进一步减小(1.998%)。

|
| 图 2 肺部菌群丰度和α多样性 A:门水平相对丰度;B:属水平相对丰度;C~E:菌群α多样性。*P<0.05。 |
结果(图 2C~图 2E)显示:4组间肺部菌群多样性总体差异不明显,GLIPR1_SMOKE组Shannon指数低于GLIPR1组(P<0.05)。
2.3 肺部菌群结构组成分析结果(图 3A)显示:PCoA可以明确划分WT组、WT_SMOKE组、GLIPR1组、GLIPR1_SMOKE组(PANOSIM=0.003),解释4组数据15.66%的变异。ANOVA分析(图 3B)显示,4组之间存在28个差异菌属。WT_SMOKE组和GLIPR1_SMOKE组小鼠肺部双歧杆菌属丰度较高,以后者更为显著。与WT组小鼠相比,类拟杆菌属、卟啉单胞菌属、毛螺菌科_(UCG-006、UCG-001)属、优杆菌属(ruminantium_group、nodatum_group)在WT_SMOKE组小鼠中占比较低,在GLIPR1_SMOKE组小鼠中占比更低。

|
| 图 3 小鼠肺部菌群β多样性 A:PCoA;B:差异菌属热图分析。 |
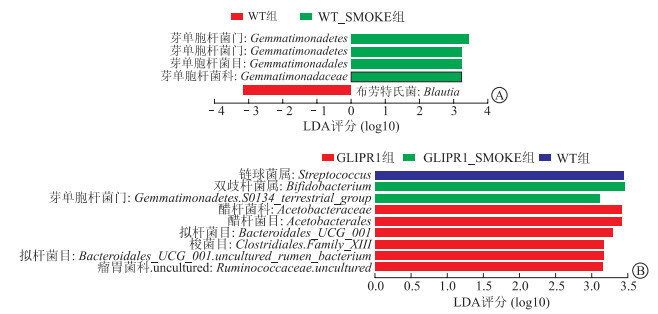
LEfSe分析(图 4)显示,与WT组相比,布劳特氏菌丰度(LDA评分=3.137 log10,P=0.006)在WT_SMOKE组占比减少0.25%,在GLIPR1组和GLIPR1_ SMOKE组中变化不明显;链球菌属(LDA评分=3.447 log10,P=0.048)在WT组丰度最高;双歧杆菌属(LDA评分=3.465 log10,P=0.042)和芽单胞杆菌门S0134_terrestrial_group(LDA评分=3.115 log10,P=0.041)在GLIPR1_SMOKE组丰度最高;醋杆菌科(LDA评分=3.429 log10,P=0.022)、醋杆菌目(LDA评分=3.418 log10,P=0.022)、拟杆菌目_UCG_001(LDA评分=3.301 log10,P=0.005)、梭菌目Family_XIII(LDA评分=3.186 log10,P=0.025)、拟杆菌目_UCG_001.uncultured_rumen_bacterium(LDA评分=3.169 log10,P=0.010)、瘤胃菌科.uncultured(LDA评分=3.147 log10,P=0.027)丰度在GLIPR1组最高。
|
| 图 4 LEfSe分析标志菌群 A:WT组与WT_SMOKE组LDA评分分布;B:GLIPR1组、GLIPR1_SMOKE组与WT组LDA评分分布。 |
课题组前期研究显示,与WT组小鼠相比,WT_SMOKE组小鼠表现明显的COPD表型,即肺部炎症细胞浸润、气道壁增厚、肺泡破坏和气道重塑,GLIPR1_SMOKE组小鼠COPD症状进一步加重;而且,GLIPR1_SMOKE组小鼠肺部蛋白累积,总细胞、中性粒细胞和巨噬细胞的浸润和IL-6分泌较WT_SMOKE组更严重[10]。本研究通过16S rRNA测序,发现4组小鼠肺部存在差异菌群。
本研究发现,小鼠肺部拟杆菌门、厚壁菌门、变形菌门和放线菌门丰度较高,且在香烟烟雾暴露和GLIPR1基因敲除后无明显变化。4组肺部菌群α多样性差异无统计学意义,这表明香烟烟雾暴露与GLIPR1基因敲除不通过改变肺部微生物群丰富度来影响COPD发展,与Sze等[13]的研究结果一致。但是,4组小鼠肺部菌群组成存在差异。野生型小鼠香烟烟雾暴露后,布劳特氏菌占比减少。布劳特氏菌具有抗炎作用[14],能利用碳水化合物合成丁酸,维护肠道黏膜完整性[15-16],可用于预防肺炎链球菌引起肺炎[17]。此外,平均乳酸杆菌属在GLIPR1_SMOKE组占比最低,仅为1.998%。另有研究[18]显示,口服乳酸杆菌能减轻流感病毒所致的小鼠肺部损伤。上述结果提示,敲除GLIPR1基因小鼠COPD症状加重与其肺部菌群组成成分的改变相关。
本研究中,4组小鼠肺部存在28个肺部菌属,其中类拟杆菌属在烟雾暴露后占比减少,GLIPR1基因敲除后减少更明显。类拟杆菌属可以产生氢、二氧化碳、甲酸、乙酸、羧酸等物质,而其他微生物可将二氧化碳和乙酸转化为甲烷,其中甲烷具有抗炎作用[19]。双歧杆菌属在烟雾暴露后占比增加,GLIPR1基因敲除后增加更明显,且是GLIPR1_SMOKE组小鼠肺部标志菌群。但是,Vieira等[20]发现,小鼠感染肺炎克雷伯菌后,口服双歧杆菌可以抑制炎症发展。出现这种差异的原因可能为:(1)COPD不同于单一肺炎克雷伯菌感染,炎症反应过程复杂;(2)GLIPR1基因敲除后,肺部的微环境改变,双歧杆菌生长受到影响。因此,双歧杆菌属在COPD中的作用仍有待进一步深入研究。芽单胞杆菌门S0134_terrestrial_group在GLIPR1_SMOKE组中占比也最高。芽单胞杆菌门S0134_terrestrial_group为革兰阴性菌,目前研究主要集中于其对环境土壤的影响,其在生物体内的作用研究较少。
综上所述,本研究发现,香烟烟雾刺激下,GLIPR1基因敲除小鼠肺部菌群的结构组成发生显著改变。其中,双歧杆菌属和芽单胞杆菌门S0134_terrestrial_group为GLIPR1基因敲除小鼠香烟烟雾暴露后肺部的标志菌群。但是本研究样本量较小,由于肺部菌群易受到环境影响,组内存在差异,未来需要扩大样本量,规范取样流程,减小误差;同时进一步分析差异菌群与炎症因子的相关性,探讨核心菌群影响COPD进展的机制。此外,肠道微生物群影响肺部微生物群,以及肺部炎症与免疫改变(这种关系被称为“肺肠轴”)[21],胃肠道-呼吸道微生物群的相互作用可能成为研究GLIPR1在COPD发展过程中作用的新思路。
利益冲突:所有作者声明不存在利益冲突。
| [1] |
CHRISTENSON S A, SMITH B M, BAFADHEL M, et al. Chronic obstructive pulmonary disease[J]. Lancet, 2022, 399(10342): 2227-2242.
[DOI]
|
| [2] |
WHO. Global Health Estimates: Life expectancy and leading causes of death and disability[EB/OL]. (2020-12-09)[2022-6-03]. https://www.who.int/data/gho/data/themes/mortality-and-global-health-estimates.
|
| [3] |
JONES B, DONOVAN C, LIU G, et al. Animal models of COPD: what do they tell us?[J]. Respirology, 2017, 22(1): 21-32.
[DOI]
|
| [4] |
DICKSON R P, ERB-DOWNWARD J R, MARTINEZ F J, et al. The microbiome and the respiratory tract[J]. Annu Rev Physiol, 2016, 78: 481-504.
[DOI]
|
| [5] |
CUTHBERTSON L, WALKER A W, OLIVER A E, et al. Lung function and microbiota diversity in cystic fibrosis[J]. Microbiome, 2020, 8(1): 45.
[DOI]
|
| [6] |
ROGERS G B, ZAIN N M, BRUCE K D, et al. A novel microbiota stratification system predicts future exacerbations in bronchiectasis[J]. Ann Am Thorac Soc, 2014, 11(4): 496-503.
[DOI]
|
| [7] |
ZHANG Q L, COX M, LIANG Z K, et al. Airway microbiota in severe asthma and relationship to asthma severity and phenotypes[J]. PLoS One, 2016, 11(4): e0152724.
[DOI]
|
| [8] |
HALDAR K, GEORGE L, WANG Z, et al. The sputum microbiome is distinct between COPD and health, independent of smoking history[J]. Respir Res, 2020, 21(1): 183.
[DOI]
|
| [9] |
MURPHY E V, ZHANG Y, ZHU W, et al. The human glioma pathogenesis-related protein is structurally related to plant pathogenesis-related proteins and its gene is expressed specifically in brain tumors[J]. Gene, 1995, 159(1): 131-135.
[DOI]
|
| [10] |
PENG W J, WU Y Y, ZHANG G, et al. GLIPR1 protects against cigarette smoke-induced airway inflammation via PLAU/EGFR signaling[J]. Int J Chron Obstruct Pulmon Dis, 2021, 16: 2817-2832.
[DOI]
|
| [11] |
HUANG Y J, NARIYA S, HARRIS J M, et al. The airway microbiome in patients with severe asthma: associations with disease features and severity[J]. J Allergy Clin Immunol, 2015, 136(4): 874-884.
[DOI]
|
| [12] |
SVERRILD A, KⅡLERICH P, BREJNROD A, et al. Eosinophilic airway inflammation in asthmatic patients is associated with an altered airway microbiome[J]. J Allergy Clin Immunol, 2017, 140(2): 407-417.e11.
[DOI]
|
| [13] |
SZE M A, DIMITRIU P A, HAYASHI S, et al. The lung tissue microbiome in chronic obstructive pulmonary disease[J]. Am J Respir Crit Care Med, 2012, 185(10): 1073-1080.
[DOI]
|
| [14] |
RASHIDI A, PELED J U, EBADI M, et al. Protective Effect of Intestinal Blautia Against Neutropenic Fever in Allogeneic Transplant Recipients[J]. Clin Infect Dis, 2022, 75(11): 1912-1920.
[DOI]
|
| [15] |
KIM S G, BECATTINI S, MOODY T U, et al. Microbiota-derived lantibiotic restores resistance against vancomycin-resistant Enterococcus[J]. Nature, 2019, 572(7771): 665-669.
[DOI]
|
| [16] |
CUI S M, GU J Y, LIU X M, et al. Lactulose significantly increased the relative abundance of Bifidobacterium and Blautia in mice feces as revealed by 16S rRNA amplicon sequencing[J]. J Sci Food Agric, 2021, 101(13): 5721-5729.
[DOI]
|
| [17] |
VERSTRAETEN S, SENCIO V, RAISE A, et al. Description of a newly isolated Blautia faecis strain and its benefit in mouse models of post-influenza secondary enteric and pulmonary infections[J]. Nutrients, 2022, 14(7): 1478.
[DOI]
|
| [18] |
KAWASE M, HE F, KUBOTA A, et al. Oral administration of lactobacilli from human intestinal tract protects mice against influenza virus infection[J]. Lett Appl Microbiol, 2010, 51(1): 6-10.
|
| [19] |
EZEJI J C, SARIKONDA D K, HOPPERTON A, et al. Parabacteroides distasonis: intriguing aerotolerant gut anaerobe with emerging antimicrobial resistance and pathogenic and probiotic roles in human health[J]. Gut Microbes, 2021, 13(1): 1922241.
[DOI]
|
| [20] |
VIEIRA A T, ROCHA V M, TAVARES L, et al. Control of Klebsiella pneumoniae pulmonary infection and immunomodulation by oral treatment with the commensal probiotic Bifidobacterium longum 5(1A)[J]. Microbes Infect, 2016, 18(3): 180-189.
[DOI]
|
| [21] |
BUDDEN K F, GELLATLY S L, WOOD D L, et al. Emerging pathogenic links between microbiota and the gut-lung axis[J]. Nat Rev Microbiol, 2017, 15(1): 55-63.
[DOI]
|
 2023, Vol. 30
2023, Vol. 30


